前回cmdstanpyを使って単回帰を解説したので、今回は重回帰分析用のコードをまとめてみる。
使用するデータ
|
x1(説明変数) |
1 |
2 |
3 |
4 |
5 |
|
x2(説明変数) |
2 |
6 |
6 |
9 |
6 |
|
x3(目的変数) |
1 |
3 |
4 |
7 |
9 |
過去の計算によると下記式が得られていた。
pythonコード
使用したpythonコードはこちら。「test.stan」の中身を解説していく。
dataの中身はstanのモデルに合わせて修正する必要があるので、それも後で修正していく。データ数が少なすぎるせいか、収束が悪かったので、深く考えずにサンプリング数を20000に増やしてみた。何かコードで間違っているところがあったらすごくうれしいです。
import os import time import numpy as np import arviz as az from matplotlib import pyplot as plt from cmdstanpy import CmdStanModel stan_file = "./test.stan" exe_file = "./test" #コンパイル if not os.path.exists(exe_file): model = CmdStanModel(stan_file=stan_file) else: model = CmdStanModel(exe_file=exe_file) data = { "N": 5, "X1": np.array([1, 2, 3, 4, 5]), "X2": np.array([2, 6, 6, 9, 6]), "Y": np.array([1, 3, 4, 7, 9]) } import multiprocessing num_cpu = multiprocessing.cpu_count() fit = model.sample( data=data, chains=4, # chain数 seed=1, # seed固定 iter_warmup=1000, # warmupの数 iter_sampling=20000, # samplingの数 parallel_chains=num_cpu, # 並列数 save_warmup=True, # warmupもCSVに保存 thin=1, # サンプリング間隔 #output_dir=output_dir, # 出力先 show_console=False, # 標準出力 show_progress=True # progress出力 ) az.style.use("arviz-darkgrid") cmdstanpy_data = az.from_cmdstanpy( posterior=fit, log_likelihood="lp__", ) ll_data = cmdstanpy_data.log_likelihood az.plot_trace(ll_data, compact=False); fit.summary()
①stanコード(X1とX2をベクトルで指定)
data {
int<lower=0> N; //データ数
vector[N] X1; //説明変数1のデータ
vector[N] X2; //説明変数2のデータ
vector[N] Y; //目的変数のデータ
}
parameters {
real b1; //X1の偏回帰係数
real b2; //X2の偏回帰係数
real a; //切片
real<lower=0> sigma; //誤差
}
model {
for (n in 1:N){
Y[n] ~ normal (X1[n]*b1 + X2[n]*b2 + a, sigma);
}
b1 ~ normal(0,1000); //b1の事前分布
b2 ~ normal(0,1000); //b2の事前分布
a ~ normal(0,1000); //aの事前分布
sigma ~ normal(0,1000); //sigmaの事前分布
}
pythonでデータを渡すところはこんな感じになる。
data = {
"N": 5,
"X1": np.array([1, 2, 3, 4, 5]),
"X2": np.array([2, 6, 6, 9, 6]),
"Y": np.array([1, 3, 4, 7, 9])
}
X1とX2に対して係数b1,b2をそれぞれ設定して計算している。

計算結果はこんな感じ。

②stanコード(Xをmatrix、bをvectorでまとめて指定)
data {
int<lower=0> N; //データ数
int<lower=0> K; //説明変数の数
matrix[N, K] X; //説明変数
vector[N] Y; //目的変数のデータ
}
parameters {
vector[K] b; //偏回帰係数
real a; //切片
real<lower=0> sigma;
}
model {
Y ~ normal(X*b + a, sigma);
a ~ normal(0, 1000);
b ~ normal(0, 1000);
sigma ~ normal(0, 1000);
}
data = {
"N": 5,
"K": 2,
"X": np.array([[1,2],
[2,6],
[3,6],
[4,9],
[5,6]]),
"Y": np.array([1, 3, 4, 7, 9])
}
matrixやvectorを使ってまとめると表記がすっきりする。pythonのdata部分はやや行列の方向に気を付けないといけないのでちょっと混乱する。

計算結果はこちら。似たような値が返ってくる
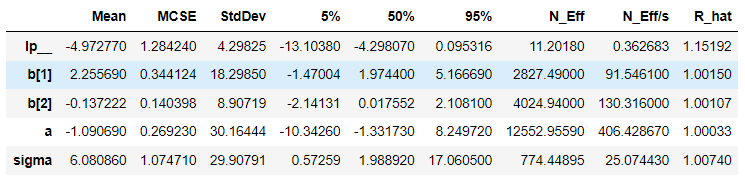
③stanコード(切片もbにまとめてみる)
よりシンプルな形にするなら、切片をまとめてしまうことも出来る。
data {
int<lower=0> N; //データ数
int<lower=0> K; //説明変数の数
matrix[N, K] X; //説明変数
vector[N] Y; //目的変数のデータ
}
parameters {
vector[K] b; //偏回帰係数+切片
real<lower=0> sigma;
}
model {
Y ~ normal(X*b, sigma);
b ~ normal(0, 1000);
sigma ~ normal(0, 1000);
}
この時はデータの指定の仕方を工夫して、最後に定数項用の列を追加する必要がある。
data = {
"N": 5,
"K": 3,
"X": np.array([[1,2,1],
[2,6,1],
[3,6,1],
[4,9,1],
[5,6,1]]),
"Y": np.array([1, 3, 4, 7, 9])
}

結果はこんな感じ。若干ずれている気もするがいいのか…?自由度が足りてなさすぎるのだろうか。
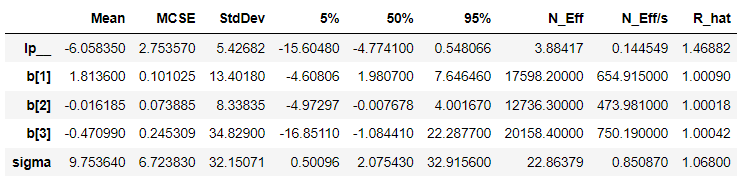
今回はこれで終わり。次回は目的変数を試してみたい。